Les réseaux métaboliques à l'échelle du génome permettent de modéliser l'ensemble des transformations chimiques connues au niveau du métabolisme d'un organisme donné. Leur comparaison à large échelle nécessite de s'affranchir des problèmes d'hétérogénéité parmi les génomes disponibles. Dans un article publié dans Genome research, des scientifiques du LBi2M, au sein de la Station biologique de Roscoff, et de l'IRISA (équipe DYLISS) proposent une approche permettant de construire des réseaux de qualité homogène permettant de les comparer dans une perspective évolutive.
Depuis une dizaine d'années, plusieurs cartes du métabolisme des algues ont été reconstruites à partir de données issues de séquençages de génomes. Lors des premières tentatives de comparaison entre ces réseaux métaboliques, les études expliquaient les différences observées entre les cartes métaboliques par l'hétérogénéité des jeux de données issus du séquençage et non par de réelles différences biologiques entre les espèces. La nouvelle approche méthodologique, implémentée dans le logiciel AuCoMe (diffusé sous licence libre), a été développée en utilisant la génomique comparative. Elle permet en s’appuyant sur les connaissances expertes contenues dans les annotations génomiques des espèces les mieux décrites, de déduire les cartes métaboliques des espèces dont les annotations des génomes sont plus rudimentaires – ou incomplètes. Des gènes manquants ont également pu être identifiés en réalisant des recherches sur les similarités des séquences entre espèces apparentées. Enfin, la méthode intègre aussi une approche pour garantir la fiabilité des prédictions en limitant les biais potentiellement induits par les spécificités d'un génome donné. Cette méthode combine des techniques d'ingénierie des connaissances, de comparaisons de séquences génomiques, et de modélisation de systèmes biologiques pour parvenir à analyser et comparer simultanément le métabolisme de plusieurs espèces eukaryotes, alors que l'état de l'art ne pouvait jusqu'à maintenant que comparer que quelques espèces, et sans pouvoir tenir compte de l'hétérogénéité des génomes.
Cette méthodologie a été appliquée pour étudier une famille d’une quarantaine de génomes d’algues très diverses : plusieurs lignées de microalgues, dont les diatomées et les cryptophytes, ainsi que des macroalgues vertes, rouges et brunes. Elle a permis de d’établir un catalogue unique de cartes métaboliques pour des dizaines d’eucaryotes marins. Les comparaisons entre ces cartes ont montré que les différences entre les métabolismes des différentes espèces étudiées sont pour l'essentiel en accord avec la phylogénie de référence, c'est à dire avec l'état actuel des connaissances sur les relations de parenté entre ces organismes. Il y a cependant certaines différences notables. En particulier, les prédictions des auteurs suggèrent que la microalgue Guillardia theta, considérée comme appartenant au groupe des cryptophytes, dont la position phylogénétique est encore discutée, a un métabolisme qui semble partager des caractéristiques communes à la fois avec les archéplastides (regroupant lignée verte et algues rouges) et les straménopiles (algues brunes et diatomées).
Cette méthode permet d'obtenir rapidement un premier aperçu du métabolisme d'un groupe d'organismes donné, ouvrant ainsi la voie à des études plus approfondies sur certaines spécificités nécessitant de faire l'objet d'études expérimentales complémentaires.
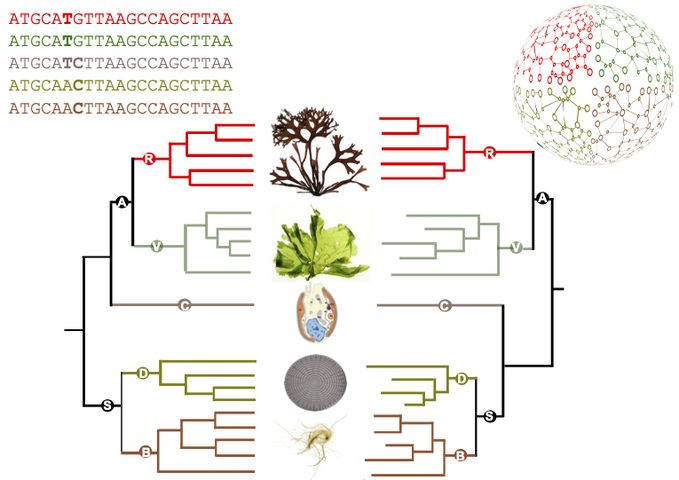
Figure : comparaison entre une phylogénie de référence générée à partir de séquences (partie gauche) et le dendrogramme métabolique correspondant, issue de la comparaison des réseaux métaboliques (partie droite). Simplifié d'après l'article.
Crédits Figure: Ryo Onuma, Neha Mishra, Shin-ya Miyagishima, George Swann (CC-BY SA 4.0), Jeanne Got (Irisa), Jonas Collén, Simon Dittami, Gabriel Markov (Station Biologique Roscoff).
Crédits vignette : George Swann (CC-BY SA 4.0), Jeanne Got (Irisa), Jonas Collén, Simon Dittami, Gabriel Markov (Station Biologique Roscoff).
Article issus du site de l'Institut INSB du CNRS
En savoir plus :
Inferring and comparing metabolism across heterogeneous sets of annotated genomes using AuCoMe
Arnaud Belcour, Jeanne Got, Méziane Aite, Ludovic Delage, Jonas Collén, Clémence Frioux, Catherine Leblanc, Simon M. Dittami, Samuel Blanquart, Gabriel V. Markov and Anne Siegel
Genome Research, juin 2023. DOI : https://doi.org/10.1101/gr.277056.122
Contacts chercheurs
Gabriel Markov, chercheur CNRS
gabriel [*] markov sb-roscoff [*] fr (gabriel[dot]markov[at]sb-roscoff[dot]fr)
sb-roscoff [*] fr (gabriel[dot]markov[at]sb-roscoff[dot]fr)
Twitter : @gabrielvmarkov
Laboratoire de Biologie Intégrative des Modèles Marins (CNRS / Sorbonne Université),
Station Biologique de Roscoff, Place Georges Teissier, 29680 Roscoff
Anne Siegel, chercheuse CNRS
anne [*] siegel irisa [*] fr (anne[dot]siegel[at]irisa[dot]fr)
irisa [*] fr (anne[dot]siegel[at]irisa[dot]fr)
Twitter : @DylissTeam
Institut de recherche en informatique et systèmes aléatoires (CNRS /Université de Rennes).
IRISA, Campus universitaire de Beaulieu - 263 Avenue du Général Leclerc - CS 74205, 35042 RENNES Cedex - France
